近日鄭春福教授與合作者在Journal of Medical Virology (JMV) 發表新文章:The binding profile of SARS-CoV-2 with HLA polymorphisms reveals critical alleles involved in immune evasion (SARS-CoV-2與HLA多態性的結合譜揭示免疫逃避的關鍵等位基因)。
由 SARS-CoV-2 引起的 COVID-19 大流行導致數百萬人死亡。由于病毒新突變和免疫逃避,SARS-CoV-2的傳播力非常迅速。然而,相關機制仍不清楚。本研究從T-CoV數據庫中獲得SARS-CoV-2變異體與HLA多態性之間的親和力數據。文獻中確定了關注變體(VOC)的流行病學數據。HLA蛋白序列從 IPD-IMGT/HLA獲得。使用AlphaFold預測蛋白質結構,并使用PrankWeb 3預測HLA分子的結合位點。結果獲得了 SARS-CoV-2 毒株與不同 HLA 蛋白的結合親和力,并鑒定了 31 個風險等位基因。 隨后的結構預測確定了這些 HLA 蛋白中的 10 個可能促進免疫逃避的活性結合位點。 特別地,還發現與 HLA I 類多態性的弱結合能力可能有助于 Omicron 的免疫逃避。總之,本研究重點關注 HLA 多態性與 SARS-CoV-2 毒株的結合親和力及其與 SARS-CoV-2 免疫逃避的關系。 研究結果對于防止 SARS-CoV-2 的免疫逃避具有重要意義,并為疫苗設計提供了新的見解。
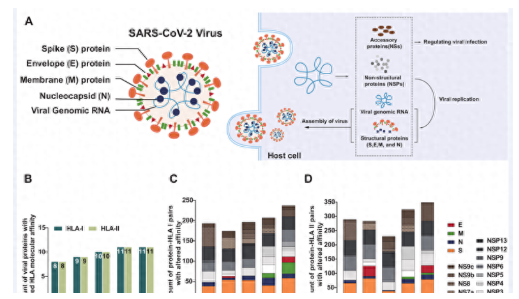
圖1. 不同SARS-COV2毒株與蛋白質結合的HLA等位基因的累積數量.
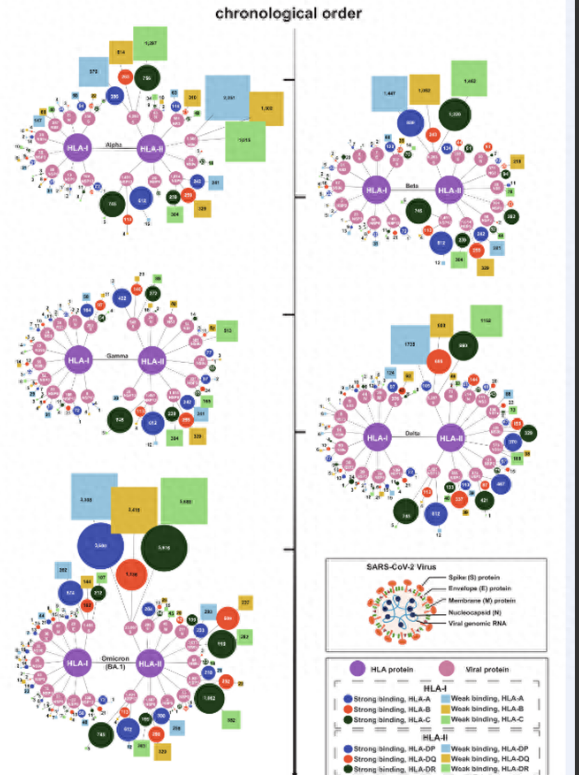
圖2. HLA分子結合SARS-COV2肽
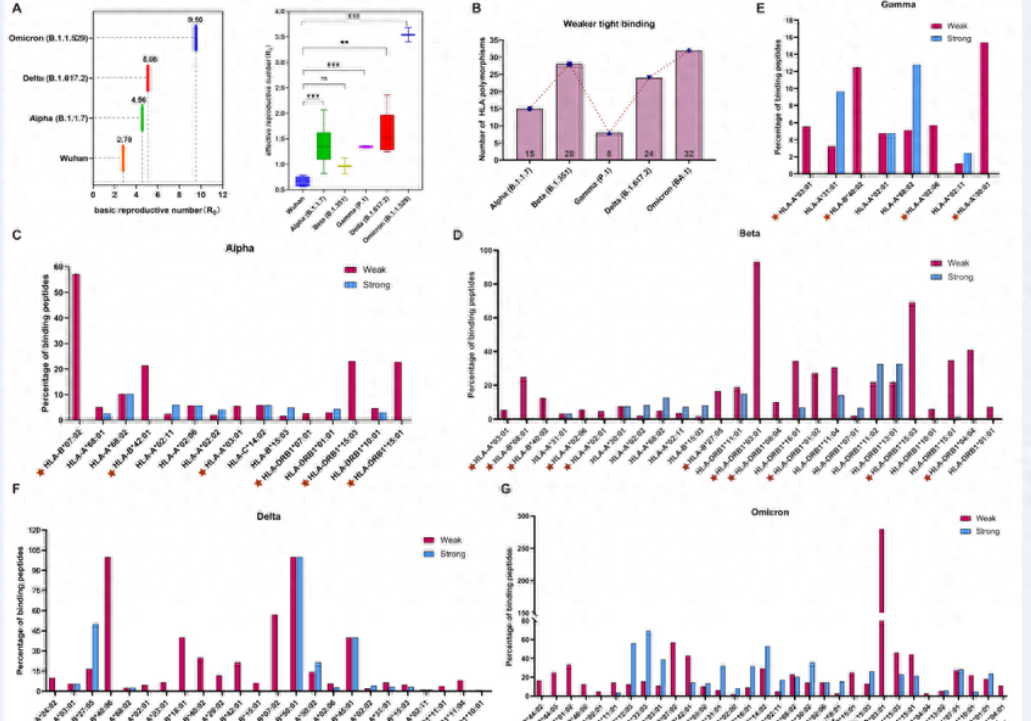
圖3. S蛋白在VOC中的結合親和力變化.

圖4. 風險等位基因種族差異的篩查和分析。

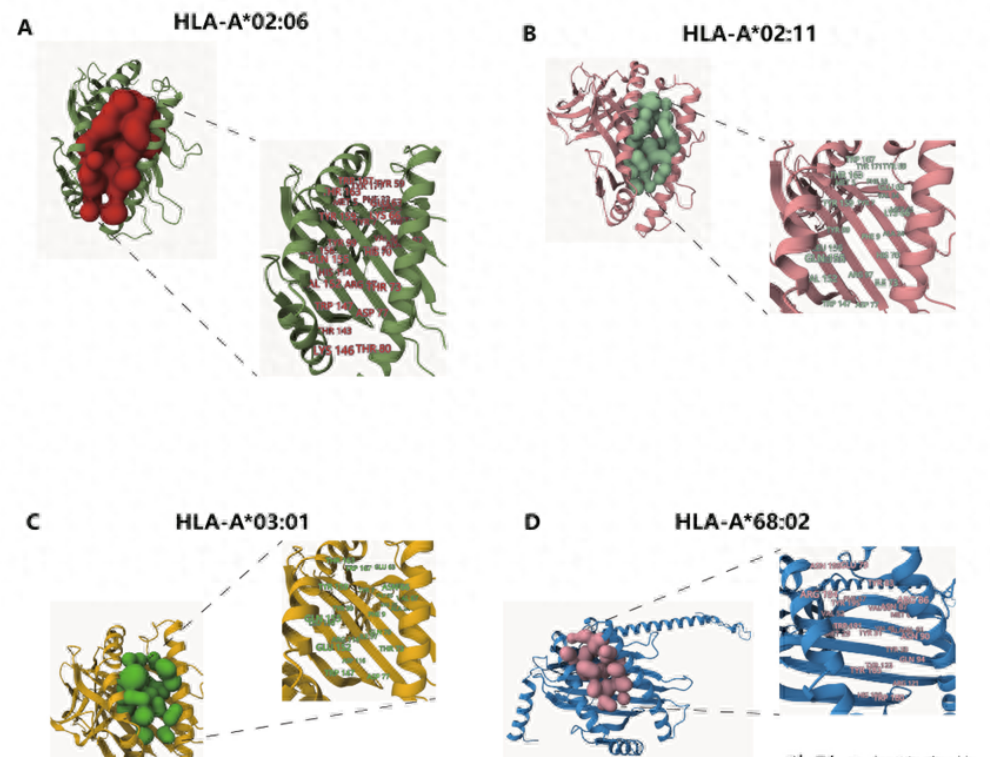
圖5. 關鍵多態性結合位點的預測.
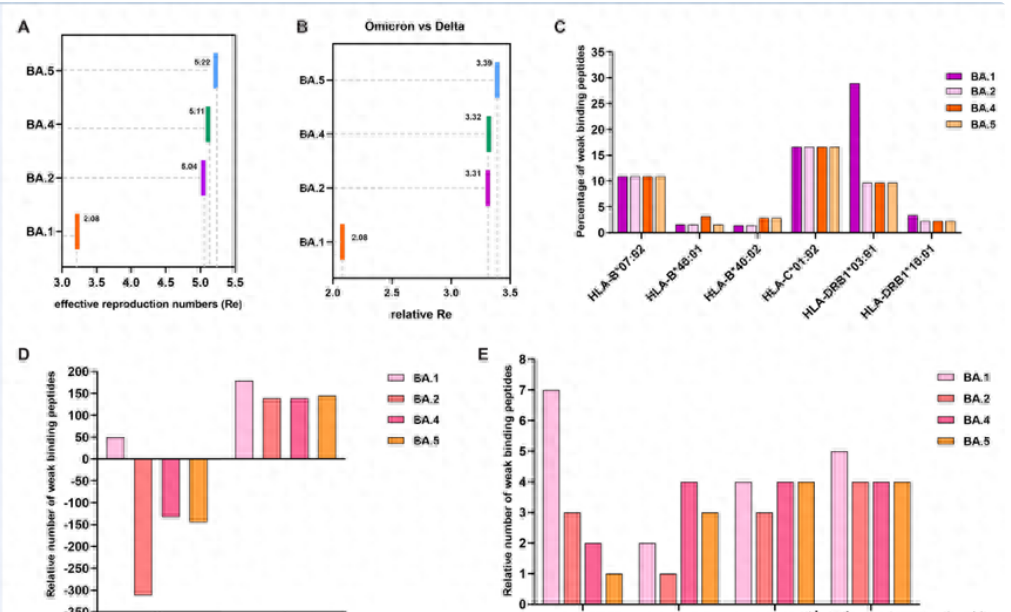
圖6. Omicro不同分支的HLA多態性及免疫逃避.




 17312606166
17312606166